[Tuto] Fabrique & comprends ton premier réseau de neurones en partant de zéro !
August 9, 2018
datascience machinelearning R tools DeepLearning AI NeuralNetworkCet article s’adresse à un public ayant des connaissances de statistiques (régression logistique) et du langage R, souhaitant s’initier au Deep Learning par une approche pratique, ou revoir les bases. Mon objectif est de partager ma compréhension avec vous, toujours en l’appuyant avec des éléments pratiques: du code et des graphiques.
Deep Learning est sans doute le buzzword de l’année dans la tech & la recherche. Dans ce qui suit, on ne s’attardera pas sur le côté théorique de la question, celui-ci méritant plus qu’un petit article comme celui-ci & ayant été largement traité par des experts.
Dans l’esprit learning by doing, adoptons plutôt une approche très pratique des choses. Dans ce qui suit, nous allons à partir de rien et en allant droit au but: construire un réseau de neurones simple. L’objectif est de mettre à nu, de déconstruire et fragmenter ce fameux réseau de neurones pour le démystifier et éliminer l’aura de complexité qui l’entoure. Nous utiliserons la célèbre régression logistique comme référence pour expliquer en quoi un réseau de neurones est différent.

Après tout, les briques de base du réseau de neurones n’ont rien de compliqué, mais les chercheurs & ingénieurs rivalisent de créativité pour en agencer des centaines voir des milliers dans des architectures ayant une fonction avancée. Ainsi aujourd’hui grâce au Deep Learning, on fait de la reconnaissance d’images (essayez ici), on traduit du texte, on prédit des évolutions de prix, on apprend à courir knacki man et on bat des champions du monde du jeu de Go ! :)
Dans ce qui suit, je m’efforce de progresser de manière claire et concrète, appuyée & illustrée par un peu de code simplifié en langage R pour le côté appliqué. Parce qu’une image vaut mieux qu’un long discours, nous mettrons l’accent sur la visualisation.
Commençons simple :
Commençons par créer des données. On se place dans le plan en deux dimensions : imaginons 250 balles de couleurs bleu & rouge disposées sur une table.
tout d’abord sous une forme simple. Deux “tas”, assez bien séparés :
# Je génère un jeu de données grâce à la fonction rnorm() de R qui prend en
# argument un nombre de points, une moyenne et un écart type de la
# distribution à générer.
nb = 125
sd = 1.5
biclust <- rbind(data.frame(x1 = rnorm(nb, 2, sd), x2 = rnorm(nb, 2.5, sd),
class = rep("Rouge", nb)), data.frame(x1 = rnorm(nb, 7.5, sd), x2 = rnorm(nb,
7.5, sd), class = rep("Bleu", nb)))
# Admirez la puissance graphique de ggplot2 :)
library(ggplot2)
theme_set(theme_minimal())
ggplot(biclust) + aes(x1, x2, colour = class) + geom_point() + labs(x = "x1",
y = "x2")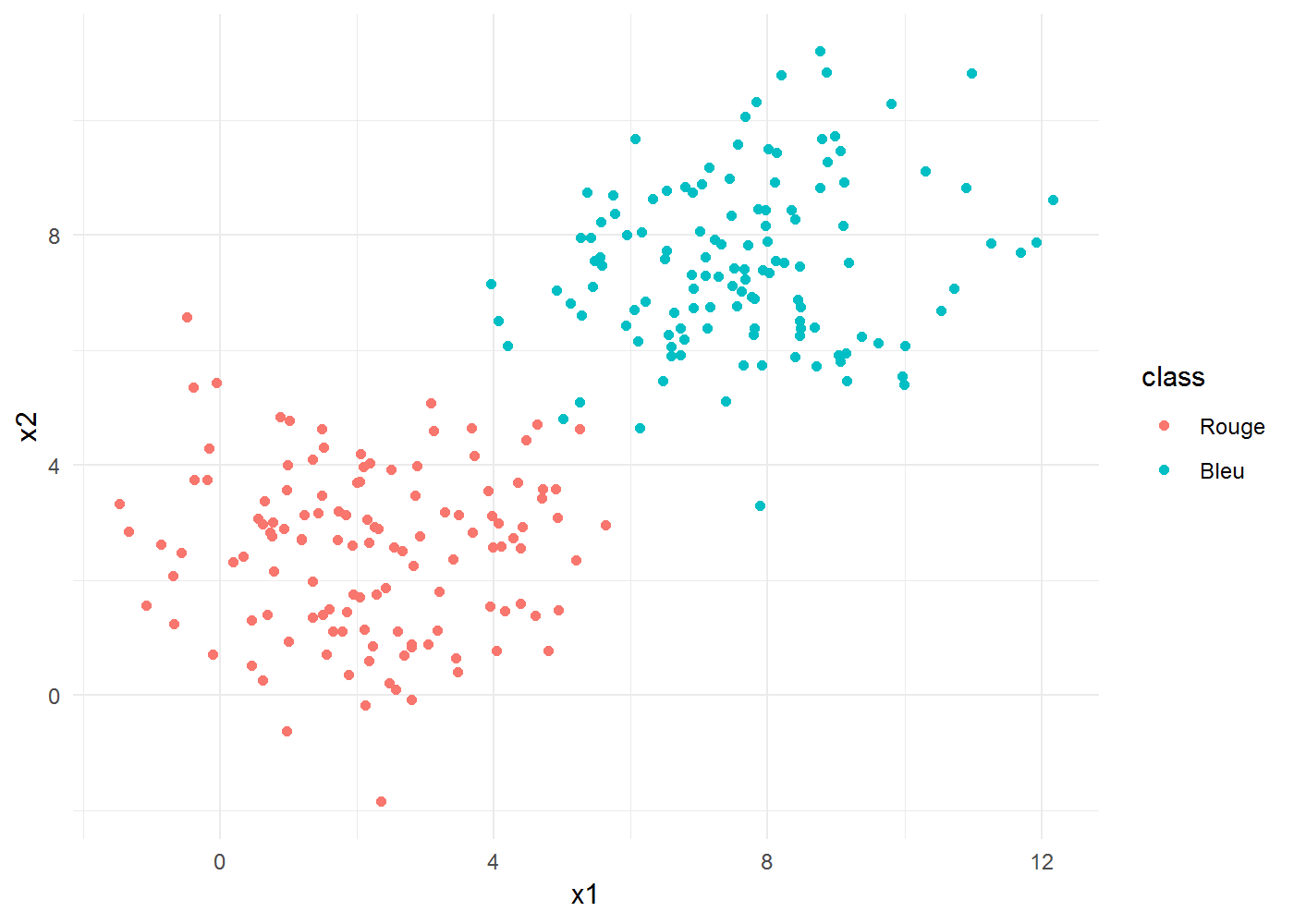
Comment pourrions-nous séparer les points rouges des bleus ? Deux choses sont à remarquer: * Nous sommes face à un problème de classification binaire, * Les points ne sont pas mélangés, il est donc possible de les séparer facilement et simplement, à l’aide d’une droite. Une fonction affine bien placée devrait faire l’affaire,
L’intuition ici est d’employer un modèle linéaire. Essayons avec une régression logistique :
Voici la manière dont notre modèle performe, que l’on peut voir à la qualité de séparation entre points bleus et rouges :
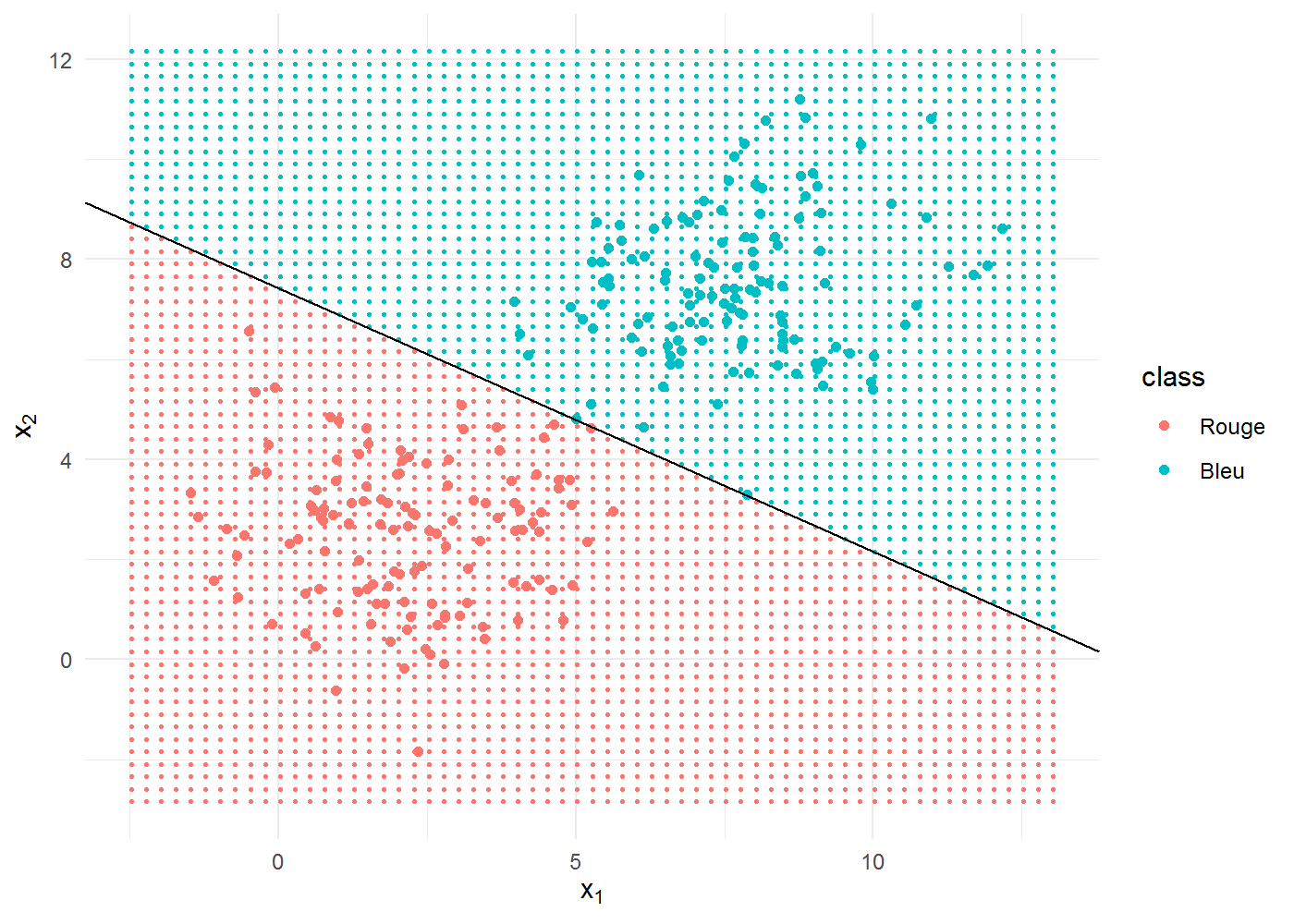
La séparation, représentée par une frontière linéaire, est quasi-parfaite. En effet, nous avons utilisé un algorithme d’apprentissage supervisé, le modèle linéaire (une droite de la forme y=a.x+b), pour résoudre un problème linéaire, c’est super, tout roule ! :)
Essayons à présent avec des données plus complexes :
Commençons par fabriquer un jeu de données de 250 points ayant une forme un peu plus exotique. Donnons lui une forme spirale, tiens !
deux_spirales <- function(N = 250, rad = 2 * pi, th0 = pi/2, labels = 0:1) {
N1 <- floor(N/2)
N2 <- N - N1
theta <- th0 + runif(N1) * rad
spiral1 <- cbind(-theta * cos(theta) + runif(N1), theta * sin(theta) + runif(N1))
spiral2 <- cbind(theta * cos(theta) + runif(N2), -theta * sin(theta) + runif(N2))
points <- rbind(spiral1, spiral2)
classes <- c(rep(0, N1), rep(1, N2))
data.frame(x1 = points[, 1], x2 = points[, 2], class = factor(classes, labels = labels))
}
set.seed(42)
spiral <- deux_spirales(labels = c("Rouge", "Bleu"))
# Graphiquement :
library(ggplot2)
theme_set(theme_minimal())
ggplot(spiral) + aes(x1, x2, colour = class) + geom_point() + labs(x = expression(x[1]),
y = expression(x[2]))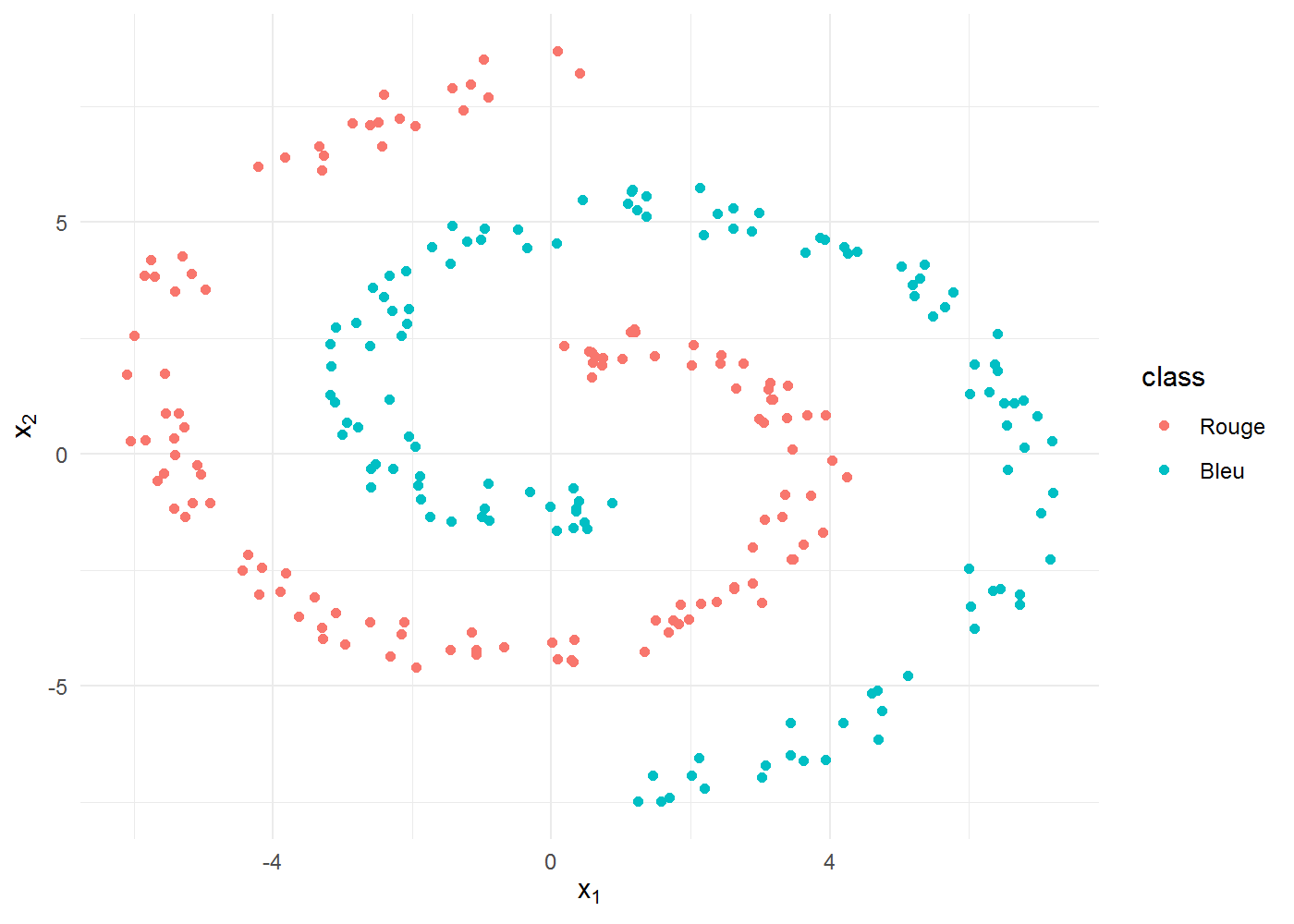
Aperçu des données :
library(DT)
datatable(head(spiral))
(et on n’oublie pas ses 5 fruits et légumes par jour !)
Commençons par ce qu’on connaît déjà
Comme précédemment, essayons la régression logistique pour voir :
logreg <- glm(class ~ x1 + x2, family = binomial, data = spiral)
correct <- sum((fitted(logreg) > 0.5) + 1 == as.integer(spiral$class))140 points sur 250 sont bien classifiés, soit une précision de 56 % seulement. Visuellement, ça donne ça :
beta <- coef(logreg)
grid <- expand.grid(x1 = seq(min(spiral$x1) - 1, max(spiral$x1) + 1, by = 0.25),
x2 = seq(min(spiral$x2) - 1, max(spiral$x2) + 1, by = 0.25))
grid$class <- factor((predict(logreg, newdata = grid) > 0) * 1, labels = c("Rouge",
"Bleu"))
ggplot(spiral) + aes(x1, x2, colour = class) + geom_point(data = grid, size = 0.5) +
geom_point() + labs(x = expression(x[1]), y = expression(x[2])) + geom_abline(intercept = -beta[1]/beta[3],
slope = -beta[2]/beta[3])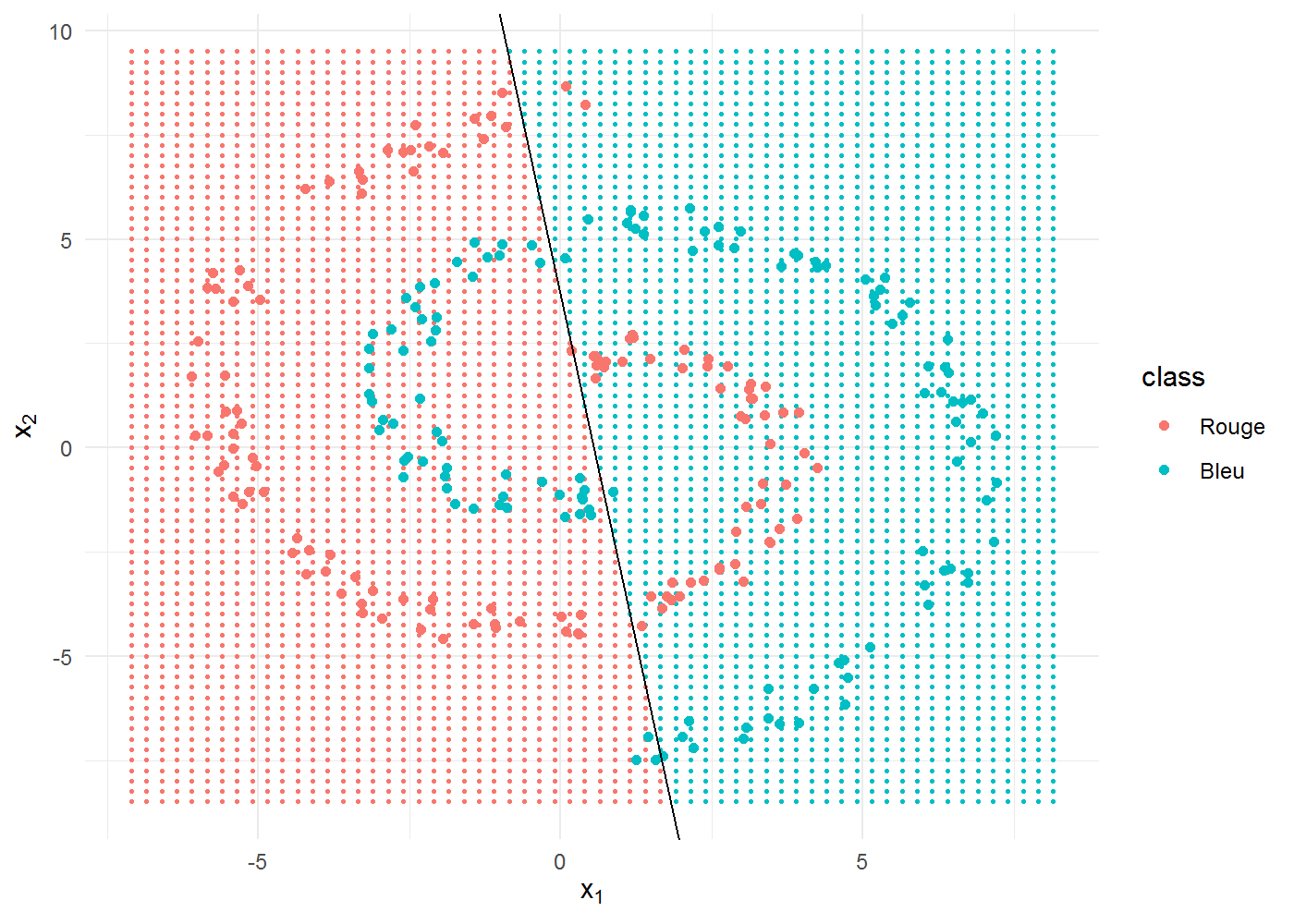
Essayons de faire mieux, pourquoi pas avec un réseau de neurones artificiels ?!
Fonctionnement général
Un réseau de neurone est un approximateur de fonction universel. C’est à dire qu’il est capable d’apprendre toutes sortes de structures de données, y compris complexes, et de reproduire (approximer) cette structure pour de nouvelles données. La brique de base du réseau de neurones est le neurone (ou perceptron).
Comme vous l’avez deviné, les réseaux de neurones artificiels sont inspirés des neurones biologiques.
Un neurone est une unité de calcul. Elle prend plusieurs valeurs en entrée et renvoie une sortie calculée, comme ceci :

Source Pollen Multimedia
Les données en entrée x1 et x2 sont combinées dans une fonction linéaire du type y=w1⋅x1+w2⋅x2+b, les W étant des coefficients donnant plus ou moins de poids à la variable selon son importance dans la sortie. Le résultat de cette fonction passe ensuite dans une fonction d’activation appelée Sigmoïde (ou fonction logistique !) qui a plein de propriétés intéressantes dont celle de placer la sortie dans l’intervalle continu [0,1]. Génial, une probabilité ! nous voulions justement prédire les chances d’un point d’être bleu plutôt que rouge (ou l’inverse, peu importe). Rappelons que lors de l’étude d’un phénomène donné, la somme des probabilité de réalisation des différentes possibilités est toujours égale à 1 ∑P(X)=1.
La puissance du perceptron se révèle lorsqu’il travaille en réseau avec un grand nombre de ses copains. L’architecture la plus fréquente, le perceptron multi-couches se présente comme suit :
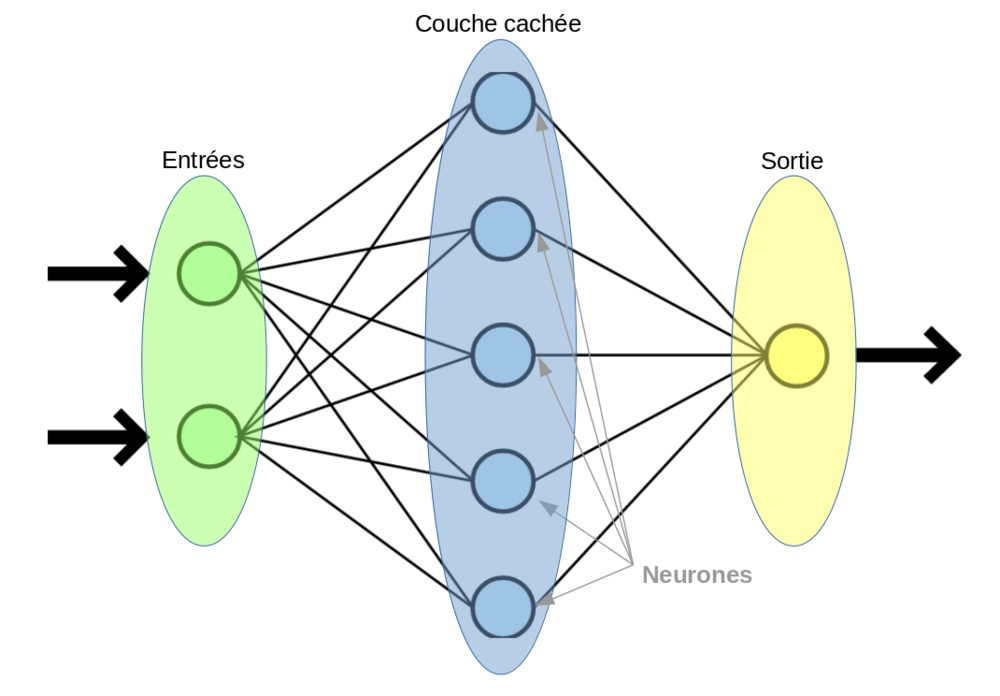
Source : http://www.natural-solutions.eu/
Ce réseau est dit dense (ou fully-connected). Tous les neurones d’une couche sont connectés à tous ceux de la prochaine.
Les couches cachées sont celles qui n’interagissent pas avec l’extérieur. Elles sont internes au réseau.
A l’attaque, codons notre premier réseau de neurones !
Codons un réseau à une couche cachée. Comme pour tout modèle, une fois que l’on a défini ses propriétés (paramètres), vient la phase d’entraînement qui consiste à montrer les données au modèle afin qu’il apprenne.
D’abord, l’architecture gloable
Nous allons coder un réseau dans lequel les données progressent dans une seule direction, toujours en avant (feed-forward). Cela veut dire que les connexions dans le réseau ne forment pas de cycle (cf. CNN), l’information se déplace dans une seule direction, de la couche d’entrée à la couche de sortie en passant par les différentes couches cachées, dans l’ordre, comme ceci :
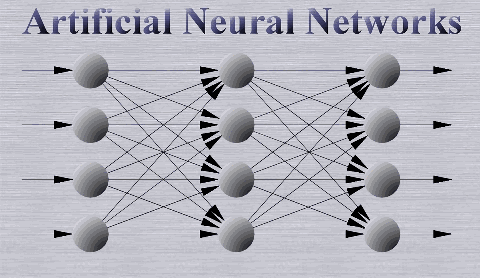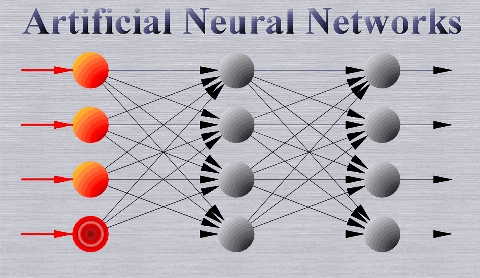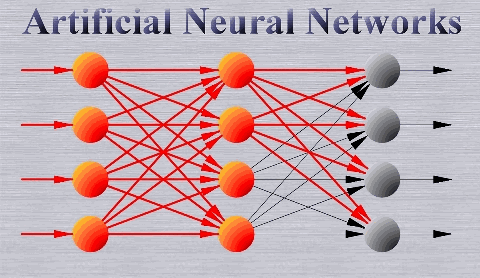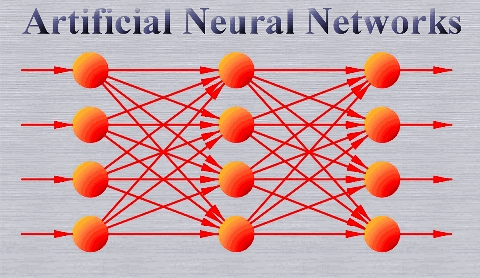
En termes de code, ça ressemble à ceci (on en profite pour faire en sorte que chaque neurone ait une Sigmoïde en fonction d’activation) :
sigmoid <- function(x) 1/(1 + exp(-x))
feedforward <- function(x, w1, w2) {
z1 <- cbind(1, x) %*% w1
h <- sigmoid(z1)
z2 <- cbind(1, h) %*% w2
list(output = sigmoid(z2), h = h)
}Ensuite, rajoutons un peu d’intelligence avec la rétro-propagation du gradient
La rétro-propagation du gradient est le cœur de la machinerie permettant d’entraîner (c’est-à-dire d’ajuster) un réseau de neurones. C’est elle qui permet progressivement de trouver les poids W optimaux qui font que notre modèle est “bon”. La rétro-propagation permet au modèle d’apprendre de ses erreurs de manière itérative et de s’améliorer au fur et à mesure qu’il ingère les données. Les poids qui contribuent à engendrer une erreur importante se verront modifiés de manière plus significative que les poids qui ont engendré une erreur marginale, de manière à minimiser l’erreur globale du réseau. L’algorithme du gradient a pour but de converger de manière itérative vers une configuration optimale des poids.
Pour passer rapidement sur les maths, quelques points à comprendre :
- Tout algorithme d’apprentissage supervisé a pour objectif d’apprendre une structure de données à partir d’une base d’apprentissage, de l’imiter. Pour ce faire, il a besoin de trouver la combinaison de poids qui permette le meilleur ajustement, la meilleure approximation des données d’apprentissage.
- Pour atteindre ce meilleur ajustement, on optimise (= minimise la valeur) d’une fonction de coût (Objectif/Loss/Cost). Cette fonction de coût mesure l’écart entre les vrais données y et les prédictions du modèle ˆy, c’est à dire l’erreur. Nous sommes bien face à un problème d’optimisation avec une fonction objectif à minimiser.
- Ce problème d’optimisation, en apparence complexe, se résout en fait efficacement grâce à l’algorithme de la descente du gradient. Le gradient est une généralisation de la notion de dérivée, dans le cas à plusieurs variables (vous trouverez ici une explication imagée et intuitive de l’algo). Dans l’espace à deux dimensions (2 variables), voici une illustration de l’algorithme. On a ici deux coefficients de régression m et b à chercher de manière à ce que notre droite apprenne les données (encart de droite). Pour ce faire, on cherche le minimum de l’erreur (encart de gauche) :

Source : www.sai-tai.com
Hop :
backpropagate <- function(x, y, y_hat, w1, w2, h, learn_rate) {
dw2 <- t(cbind(1, h)) %*% (y_hat - y)
dh <- (y_hat - y) %*% t(w2[-1, , drop = FALSE])
dw1 <- t(cbind(1, x)) %*% (h * (1 - h) * dh)
w1 <- w1 - learn_rate * dw1
w2 <- w2 - learn_rate * dw2
list(w1 = w1, w2 = w2)
}Rassemblons tout ça et testons !
On définit donc une fonction train(); permettant d’entraîner notre premier réseau de neurones, avec une architecture feed-forward et utilisant la rétro-propagation pour trouver les meilleurs paramètres. Les paramètres sont :
- x : matrice comprenant les données d’apprentissage (x1 et x2)
- y : vecteur contenant la variable réponse (label : “Rouge” ou “Bleu”)
- hidden : nombre de nœuds cachés de la couche cachée
- learn_rate : learning rate ou taux d’apprentissage. Ce paramètre contrôle le pas avec lequel s’effectue la descente du gradient. Plus il est grand, et plus la descente se fait à grands pas et donc rapidement (en théorie), mais on prend le risque de “sauter” le minimum de la fonction. Prenons donc une petite valeur (par défaut 0.01)
- iterations : nombre d’itérations (une itération correspond à un passage des données dans le réseau suivi d’une mise à jour des coefficients par rétro-propagation)
train <- function(x, y, hidden = 5, learn_rate = 0.01, iterations = 10000) {
d <- ncol(x) + 1
w1 <- matrix(rnorm(d * hidden), d, hidden)
w2 <- as.matrix(rnorm(hidden + 1))
for (i in 1:iterations) {
ff <- feedforward(x, w1, w2)
bp <- backpropagate(x, y, y_hat = ff$output, w1, w2, h = ff$h, learn_rate = learn_rate)
w1 <- bp$w1
w2 <- bp$w2
}
list(output = ff$output, w1 = w1, w2 = w2)
}Essayons avec le jeu de données spirale avec une couche cachée composée de 5 nœuds et 100000 itérations :
En termes de performance de prédiction, on est à 88.4 %, ce qui est vraiment pas mal avec des données complexes à saisir et un entrainement très rapide (une quinzaine de secondes sur un MacBook Air).
Graphiquement, il est intéressant d’observer la forme de la frontière de décision, non linéaire mais assez anguleuse quand même. On commence à percevoir la puissance d’apprentissage des réseaux de neurones :
ff_grid <- feedforward(x = data.matrix(grid[, c("x1", "x2")]), w1 = nnet5$w1,
w2 = nnet5$w2)
grid$class <- factor((ff_grid$output > 0.5) * 1, labels = levels(spiral$class))
ggplot(spiral) + aes(x1, x2, colour = class) + geom_point(data = grid, size = 0.5) +
geom_point() + labs(x = expression(x[1]), y = expression(x[2]))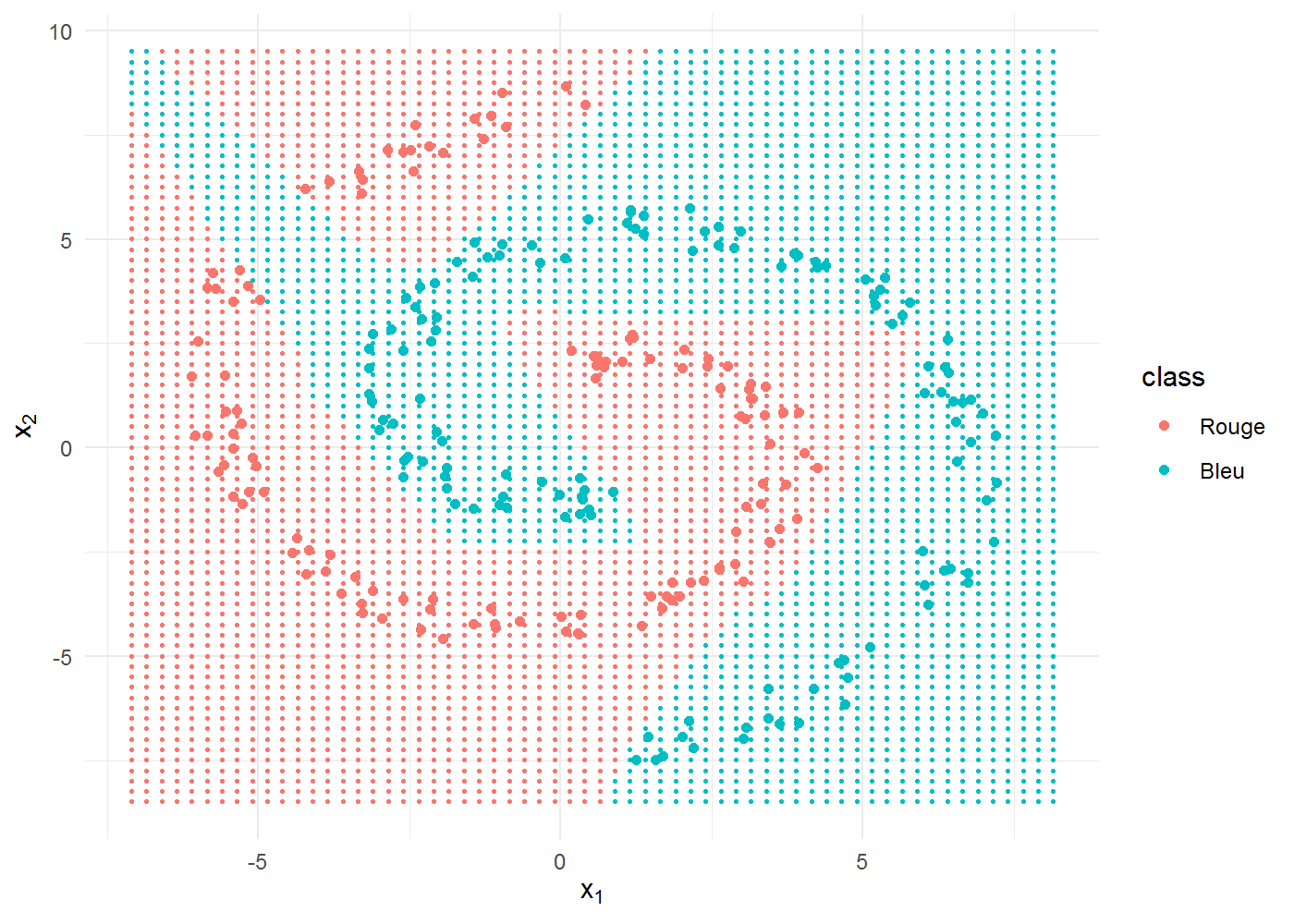
On entraîne à présent un réseau avec une couche cachée plus grande, constituée de 30 nœuds :
nnet30 <- train(x, y, hidden = 30, iterations = 1e+05)
ff_grid <- feedforward(x = data.matrix(grid[, c("x1", "x2")]), w1 = nnet30$w1,
w2 = nnet30$w2)
grid$class <- factor((ff_grid$output > 0.5) * 1, labels = levels(spiral$class))
ggplot(spiral) + aes(x1, x2, colour = class) + geom_point(data = grid, size = 0.5) +
geom_point() + labs(x = expression(x[1]), y = expression(x[2]))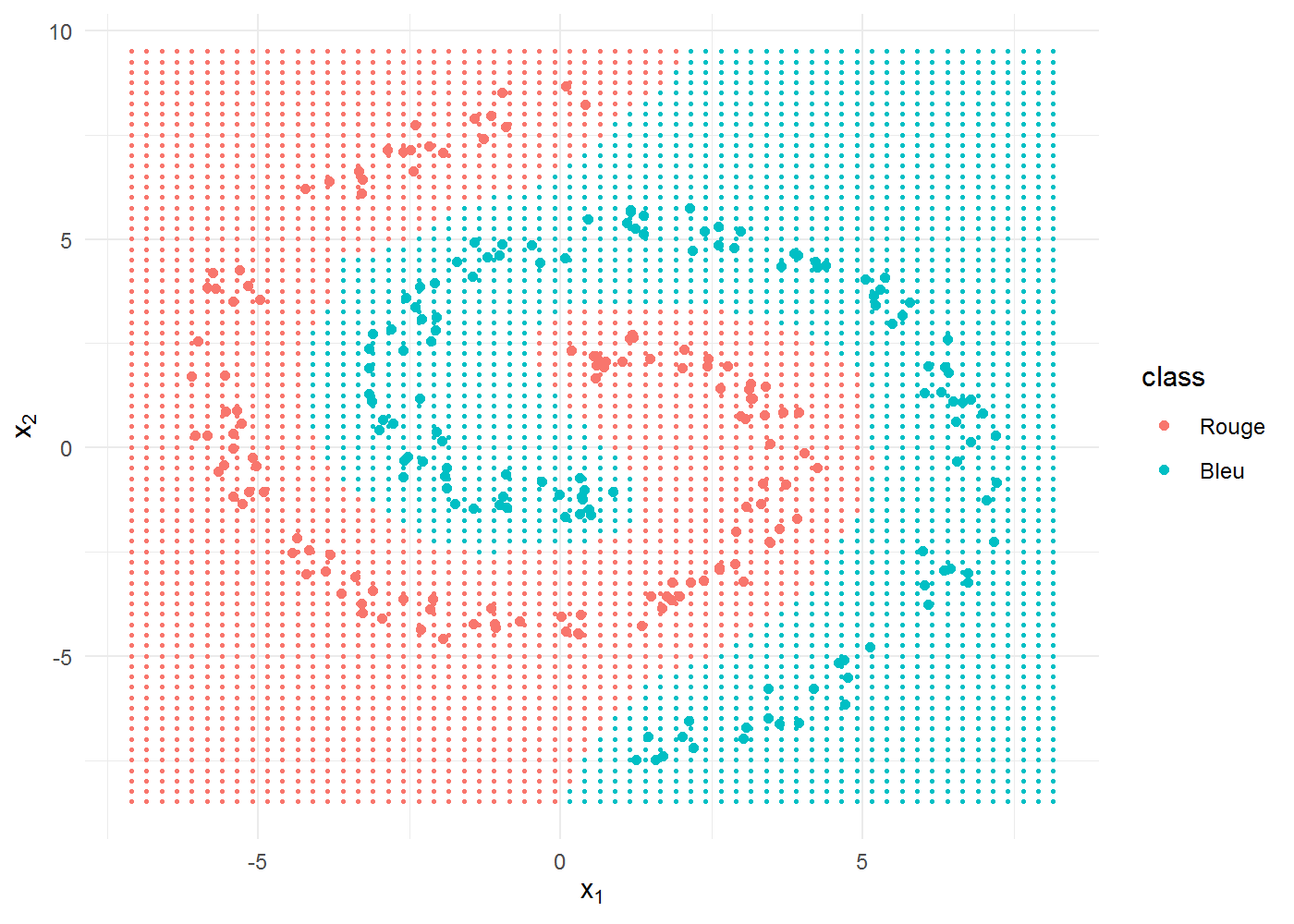
La frontière de décision est totalement arrondie, parfaitement adaptée à nos données. La performance est de … 100% !!!
Testons à présent le cas avec un seul neurone dans la couche cachée :
nnet1 <- train(x, y, hidden = 1, iterations = 1e+05)Nous voilà de retour au cas de la régression linéaire, une frontière de décision linéaire et une performance médiocre :
ff_grid <- feedforward(x = data.matrix(grid[, c("x1", "x2")]), w1 = nnet1$w1,
w2 = nnet1$w2)
grid$class <- factor((ff_grid$output > 0.5) * 1, labels = levels(spiral$class))
ggplot(spiral) + aes(x1, x2, colour = class) + geom_point(data = grid, size = 0.5) +
geom_point() + labs(x = expression(x[1]), y = expression(x[2]))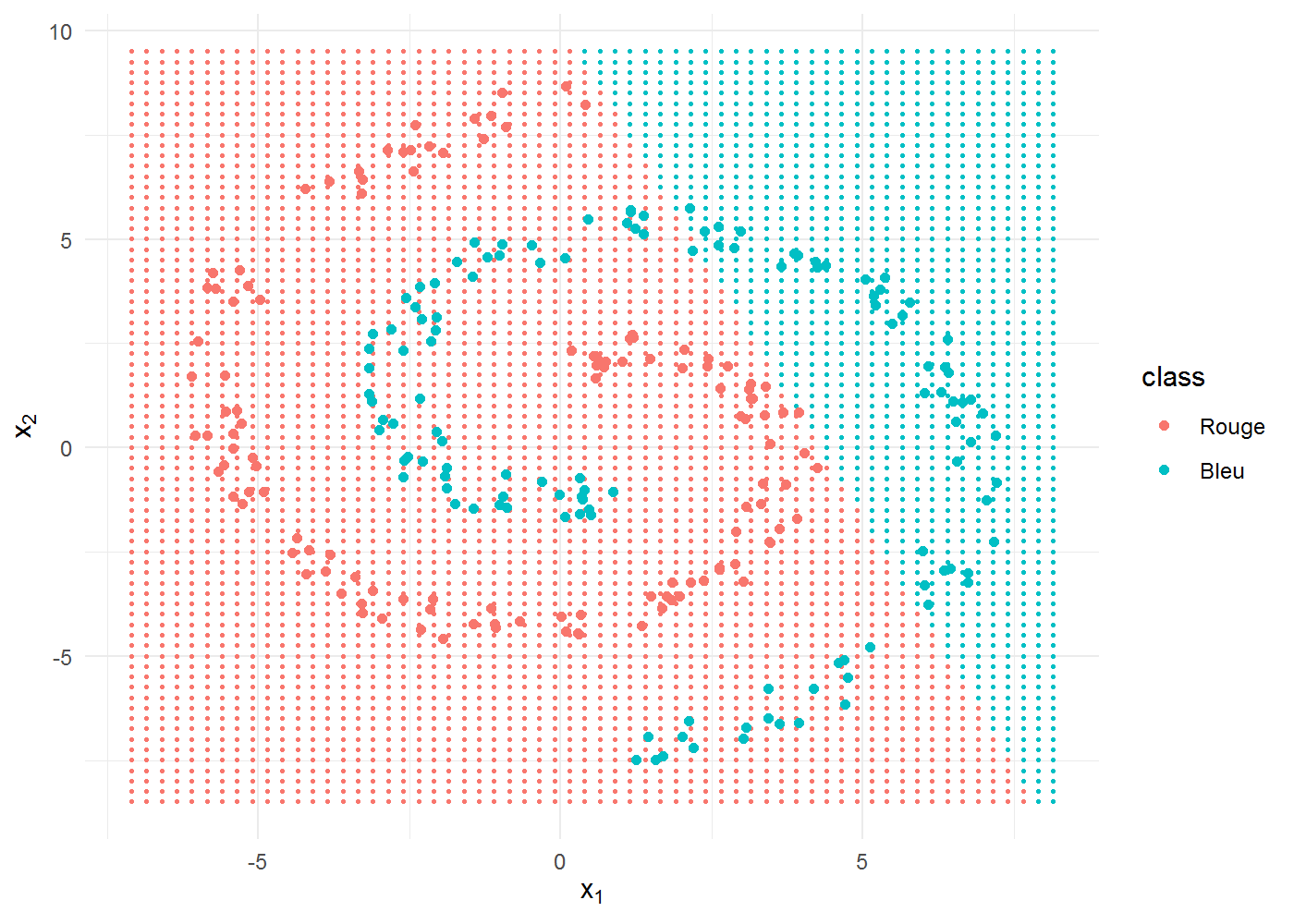
Conclusion :
Les réseaux de neurones sont une classe d’algorithmes d’apprentissage supervisé capables de résoudre des problèmes complexes. Malgré une réputation de boîte noire, un réseau de neurones est à peu de choses près un ensemble de fonctions linéaires inter-connectées. C’est justement de cette collaboration entre fonctions simples, agrémentées d’une pincée de non-linéarité que naît la puissance d’apprentissage des réseaux de neurones.
Il existe des architectures de réseaux plus complexes que celle que nous avons vue, capables de tâches très avancées comme la synthèse vocale ou la traduction de texte, et qui sont déjà employés dans énormément d’applications de notre quotidien. En voici un aperçu.

Reconnaissance d’objets

Terminator
Bien sûr, le Deep Learning “moderne” utilise des librairies logicielles spécialisées très efficaces comme PyTorch ou TensorFlow.
Côté hardware, vu le caractère hautement itératif et le grand besoin en puissance de calcul, on utilise très souvent des processeurs graphiques ou GPU (les mêmes que ceux employés pour faire tourner des jeux vidéos) plutôt que le micro-processeur de son laptop. On obtient ainsi une grande accélération de l’entraînement des modèles.
Un super article sur les avancées récentes (fin 2017 mais pertinent!) est par ici !
Pour aller plus loin, je conseille vivement de suivre le cours mis en ligne par l’équipe d’Andrew Ng sur Coursera et qui s’appelle Neural Networks and Deep Learning. Il est particulièrement adapté aux débutants car il part véritablement de zéro.
Sinon, pour aller plus loin en Deep Learning avec R, il existe le package neuralnet

En prime, petit panorama des frameworks de Deep Learning actuellement actifs. Tensorflow (Google) est le plus employé et dispose d’une large communauté, mais PyTorch (Facebook) monte très fort. Attention, il y a une petite imprécision dans cette image, Keras n’est pas un framework (back-end) de Deep Learning mais plutôt une interface de haut niveau simplifiée vers différents back-end:
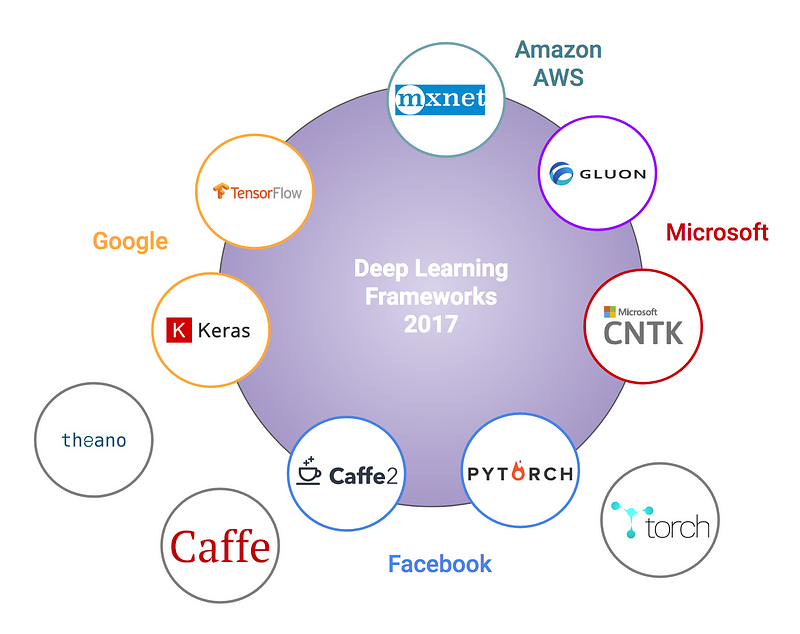
Choose wisely.
Sources :
- Google Playground
- Building a neural network from scratch in R par David Selby
- Implementing a Neural Network from Scratch in Python – An Introduction
N’hésitez pas à commenter, ceci est une première version largement améliorable et sûrement incomplète